Efectos del Entrenamiento de Fuerza de Alta Intensidad sobre la Expresión del ARNm de la Isoforma de Miosina Pesada en Estado Estable
Darryn S Willoughby1 y Stephen Pelsue2
1Department of Kinesiology, Texas Christian University, Fort Worth, TX.
2Department of Applied Medical Sciences, University of Southern Maine, Portland, ME.
Artículo publicado en el journal PubliCE, Volumen 0 del año 2000.
Publicado 19 de mayo de 2003
Resumen
Palabras clave: fuerza, traducción, pre-traducción, TR-PCR
INTRODUCCIÓN
El músculo esquelético humano es un tejido altamente adaptable que demuestra comúnmente una plasticidad significativa en respuesta a varios tipos de ejercicio. Esta plasticidad es bien ilustrada por la diversidad de isoformas (Tipo I, IIa y IIb) de los genes de la miosina de cadena pesada (MHC) sarcomérica (1). Todas las isoformas de MHC están codificadas por una familia multigenética (2) cuidadosamente conservada de distintos genes que son expresados en una forma específica de acuerdo al tejido y regulada por el desarrollo (1, 3). La diversidad de estas isoformas es incrementada en algunas instancias por una división de ARN alternativo de genes de MHC individuales, de este modo, los cambios en la actividad muscular por el entrenamiento de la fuerza alteran la expresión del gen de MHC y el fenotipo y afectan los diferentes tipos de fibras musculares de un modo característico (4). La adaptabilidad del músculo esquelético parece residir en la habilidad de las fibras musculares para transcribir diferentes isoformas de la proteína MHC, cada una de las cuales tiene características funcionales específicas (5), relativas a la contracción muscular. Consecuentemente, el polimorfismo de la MHC juega un rol principal en la adaptabilidad y los esfuerzos contráctiles de las fibras musculares necesarios para varios tipos de ejercicio.
El entrenamiento de la fuerza de alta intensidad en humanos es caracterizado por incrementos en la fuerza e hipertrofia muscular (6, 7, 8). Frecuentemente se piensa que estas adaptaciones, inducidas por el entrenamiento, son el resultado de incrementos en el contenido de proteínas miofibrilares (9, 10) y de las proteínas de la isoforma de MHC (6, 7, 8), lo que se presume que ocurre a partir de una expresión incrementada del ARNm de la isoforma de MHC. Sin embargo, en el tiempo presente, poco es conocido acerca de las características de expresión del gen MHC en respuesta al entrenamiento de la fuerza de alta intensidad en humanos.
Los incrementos en la abundancia del contenido de proteínas de la isoforma de MHC contribuyen a incrementar el contenido de proteínas miofibrilares y podrían ser el resultado de: 1. una transcripción incrementada del ARNm de la isoforma de MHC, y por esto resultando en más moléculas de ARNm que son traducidas o 2. una tasa incrementada de traducción de cada molécula de ARNm (11). También ha sido concluido que la síntesis de proteínas musculares incrementada puede también ser causada por eventos moleculares que ocurren post-traducción (9). Nosotros categorizamos el término pre-traducción al los eventos que alteran la abundancia de ARNm. Inicialmente, la abundancia es la suma algebraica de la transcripción, procesamiento y estabilidad del ARNm. El término traducción indica cambios en la síntesis de proteínas en relación a la actividad del ARNm, mientras que post-traducción es indicativo de modificación de proteínas (i.e., fosforilación, proteólisis, etc.).
En el tiempo presente, existen pocos estudios que hayan investigado la expresión del ARNm de la isoforma de MHC en respuesta al entrenamiento de la fuerza de alta intensidad en humanos. La mayoría de los estudios de entrenamiento de la fuerza que investigaron la respuesta de la MHC al entrenamiento en humanos, se han focalizado sobre las isoformas de proteínas. Como resultado, está bien establecido que las isoformas de las proteínas MHC experimentan transiciones inducidas por el entrenamiento a un fenotipo más bajo (IIb→IIa→I). En los humanos, ha sido demostrado que luego de 3 semanas de inmovilización, 12 semanas de entrenamiento de la fuerza de los extensores de la rodilla no produjeron cambios significativos en el ARNm de las 3 isoformas de MHC (12). Similarmente, también ha sido demostrado en humanos ancianos, que 7 días de entrenamiento de la fuerza de los extensores de la rodilla al 80 % del máximo no produjeron cambios significativos en los niveles de ARNm de la isoforma de MHC (11). Usando un método cualitativo de la técnica de reacción en cadena de la polimerasa (PCR), nosotros hemos demostrado (13) recientemente que la expresión relativa del ARNm de la MHC de Tipo I y IIa en ancianos, puede incrementarse después de entrenamiento de fuerza de alta intensidad, como es similar a los cambios respectivos en la expresión de las proteínas MHC, observados en otros estudios (6, 7, 8). Sin embargo, la mayor debilidad asociada con la técnica PCR cualitativa es que la misma solo proporciona la habilidad de detectar la presencia o ausencia del ARNm blanco (target ARNm), como mucho, el mismo puede solo ser pseudo-cuantificado basándose en la normalización con un patrón de control externo. De este modo, con el objeto de determinar mejor la expresión del ARNm de la isoforma de MHC usando PCR, debería ser usado un método cuantitativo.
Con respecto al procedimiento PCR usado, debería ser destacado que en estado estable, la expresión de ARNm usualmente iguala los patrones de expresión de la proteína MHC, de este modo, es asumido que la expresión de la MHC es regulada principalmente al nivel pre-traducción (14). Sin embargo, la sobrecarga mecánica como el entrenamiento de la fuerza puede realmente crear un desequilibrio entre la expresión relativa de ARNm de la MHC y las proteínas, sugiriendo que un aumento de la regulación de los genes de la isoforma de MHC como resultado del ejercicio puede no estar directamente correlacionado a la síntesis de las proteínas MHC, respectivas. Sin embargo, actualmente hay una cantidad limitada de datos disponibles sobre la expresión de ARNm de la isoforma de MHC después del entrenamiento de la fuerza de alta intensidad. Como resultado de esto, el propósito de este estudio fue: 1. determinar el nivel de abundancia de ARNm de la isoforma de MHC en estado estable y las características de la expresión del gen de MHC en respuesta al entrenamiento de fuerza de alta intensidad y 2. comparar la expresión del ARNm de MHC después del entrenamiento, usando un método de PCR cuantitativo y cualitativo.
MATERIALES Y MÉTODOS
Sujetos
12 sujetos desentrenados con una edad promedio de 19,88 ± 0,53 años, una talla de 180,13 ± 3,00 cm, y una masa corporal de 74, 63 ± 7,92 kg se ofrecieron para participar en el estudio y fueron asignados al azar a un grupo control [CON, (n =6)], el cual no realizó entrenamiento de la fuerza o a un grupo de entrenamiento de la fuerza [STR, (n = 6)]. A los sujetos con contraindicaciones para el ejercicio como fue establecido por el American Collage of Sports Medicine (Colegio Americano de Medicina del Deporte) (ACSM) y/o que hallan realizado entrenamiento de la fuerza consistente 6 meses antes del estudio, no les fue permitido participar en el estudio. Todos los sujetos elegidos firmaron documentos de informe de consentimiento aprobados por la universidad, y fue dada una aprobación por el Comité de Revisión Institucional para Utilización de Sujetos Humanos. Adicionalmente, todos los procedimientos experimentales implicados en el estudio se ajustaron a las consideraciones éticas del Código de Heslsinki. Se le explico a los sujetos el propósito del programa de entrenamiento, el protocolo a seguir, y los procedimientos experimentales a ser usados. Los sujetos fueron instruidos para mantener sus regímenes dietarios normales y para no consumir ningún tipo de suplemento deportivo (e.g., monohidrato de creatina, proteínas, etc.).
Biopsias Musculares
Fueron obtenidas biopsias musculares percutaneas (20-30 mg) antes y después del período de entrenamiento de 8 semanas para cada sujeto. Las biopsias iniciales fueron obtenidas 1 semana antes de la iniciación del entrenamiento (para permitir una cicatrización adecuada) y las biopsias siguientes fueron completadas dentro de una hora después de la última sesión de entrenamiento. Las muestras musculares extraídas bajo anestesia local (2% de xilocaina), fueron tomadas de la porción media del músculo vasto lateral derecho al punto medio entre la rótula y el trocánter mayor del fémur a una profundidad de 1 a 2 cm. Para las biopsias post-entrenamiento, se intento extraer el tejido de aproximadamente el mismo lugar, usando la cicatriz de la biopsia anterior y las marcas de profundidad en al aguja. Una incisión sucesiva fue hecha aproximadamente a 5 cm de la anterior, del plano medial al lateral (13). Los especimenes musculares fueron congelados en nitrógeno líquido para análisis posteriores.
Evaluación de la Fuerza y Determinación del Volumen del Muslo
Antes y después del período de entrenamiento de 8 semanas, ambos grupos fueron sujetos a sesiones de evaluación en las cuales fue determinada la fuerza máxima del tren inferior de cada sujeto [una repetición máxima (1 RM)], usando una máquina de prensa bilateral de piernas (Cybex, Owatonna, MN). Debido a diferencias en la fuerza muscular absoluta y en la masa corporal, entre los grupos al comienzo del estudio, la fuerza absoluta de 1 RM de cada sujeto fue dividida por su peso corporal para obtener una medición relativa de la fuerza. La fuerza relativa fue usada como una variable de criterio debido a que la misma corrige variaciones en el peso corporal entre los sujetos, y por esto provee una estimación de la fuerza más exacta (15, 16).
El volumen del muslo (m³) fue estimado antes y después del estudio a partir de una ecuación y de lineamientos previamente establecidos (17). Teniendo en cuenta las mediciones de longitud, circunferencia, y grosor de los pliegues cutáneos del muslo derecho de cada sujeto. La medición fue realizada en la posición supina y siempre antes de realizar ejercicio para eliminar la influencia de una posible inflamación muscular, inducida por el ejercicio.
Protocolo de Entrenamiento
Los principios del entrenamiento de sobrecarga y resistencia progresiva fueron incorporados en el programa de entrenamiento de la fuerza basándose en lineamientos previamente establecidos (13, 15, 16). Además de los pre-test de 1 RM, la RM fue evaluada cada 2 semanas para evaluar la fuerza muscular continuamente, con el objeto de que los ajustes en la rutina de entrenamiento pudieran acoplarse a los incrementos de la fuerza y para asegurar que los sujetos continuaran entrenando al 85-95 % de su 1 RM, basándose en el continum de repeticiones y en las guías previamente establecidas (18, 19).
Las sesiones de entrenamiento se realizaban 3 días/semana, los días lunes, miércoles y viernes y estaban conformadas por sesiones de 30 minutos (excluyendo la entrada en calor y la vuelta a la calma). El diseño y la intensidad del protocolo de entrenamiento implicaban 3 series de 6-8 repeticiones al 85-95 % de 1 RM. Fue requerido un período de descanso de 90 segundos entre cada serie y cada ejercicio para ayudar a contrarrestar la fatiga (15). Para la entrada en calor y la vuelta a la calma, los trabajos empezaban y terminaban con 10 minutos de ejercicios de flexibilidad combinados con ejercicios calinestésicos. Los miembros del grupo CON no participaron en ningún entrenamiento de la fuerza durante el curso del estudio (otro que las evaluaciones de fuerza pre y post-entrenamieto). Las sesiones de entrenamiento perdidas eran realizadas los martes y los jueves y se les informo a los sujetos que perder 3 sesiones resultaría en la descalificación del estudio.
Aislamiento del ARN total
El ARN celular total fue extraído de la homogeinización de las muestras de las biopsias con una solución monofásica de fenol e isocinato de guanidina (13, 20, 21). Específicamente, las muestras fueron homogeneizadas e incubadas con el agente TRI (Sigma Chemical Co., St. Louis, MO). El ERN fue precipitado con isopropanol, lavado con 70 % de etanol y resuspendido en H2Od. La concentración de ARN fue determinada por medio de densidad óptica (OD) a 260 nm (usando un equivalente OD260 a 40 μg/μL), y la concentración final fue ajustada a 1 μg/μL (5, 12, 20). Las alícuotas (5 μg/μL) de las muestras de ARN total fueron luego separadas con bromuro de etidio, y controladas bajo una luz ultravioleta para verificar la integridad del ARN y la ausencia de degradación de ARN. Este procedimiento dio como resultado ARN no degradado, libre de ADN y proteínas, como fue indicado por las prominentes bandas de ARN ribosomal 28S y 18S (Figura 1), así como un índice OD260/OD280 de aproximadamente 2.0 (5, 13, 21). Las muestras de ARN fueron almacenadas a -70°C hasta los análisis posteriores.
Transcripción Invertida y Síntesis de ADNc
Dos microgramos de ARN del músculo esquelético fueron transcriptos de forma inversa para sintetizar ADNc (21). Una mezcla de la reacción de transcripción invertida (RT) [(2 μg) de ARN celular, amortigudor de transcripción invertida 10x (20 mM Tris-HCL, ph 8,3; 50 mM KCl; 2.5 mM MgCl2; 100 μg de de albúmina sérica bovina/ml), una mezcla NTPd, conteniendo 0.2 mM cada ATPd, CTPd, GTPd, y TTPd, 0.8 μM MgCl2, 1.0 U/μl de RNasinr (inhibidor de la ribonucleasa), 0.5 μg/μL de primer oligo (dT)15, y 25 U/μg de la enzima AMV transcriptasa invertida (Promega, Madison, WI)] fue incubada a 42 ºC por 60 minutos, calentada a 95 ºC por 10 minutos, y luego rápidamente enfriada en hielo. La concentración inicial fue estandarizada por medio del ajuste de las reacciones RT para todas las muestras a 200 ng, antes de la amplificación PCR (5).
Primeres Oligonucleotídicos para PCR cualitativa y cuantitativa
El 5’-oligonucleótido de cada amplificación de reacción en cadena de la polimerasa (PCR) fue concebido a partir de una región altamente conservada en todos los genes humanos conocidos de MHC, aproximadamente 600 pares de bases (bp) en dirección ascendente a partir del cordón de finalización. Las tres isoformas de MHC adultas (Tipo I, IIa y IIb) son idénticas en esta región, lo que nos permitió usar el mismo primer en dirección ascendente “en un sentido común” con la siguiente secuencia: 5'-GCCAAGAAGGCCATCAC-3' (13, 21, 22). Los primers 3’-oligonucleotidicos en antisentido usados en las reacciones PCR fueron concebidos a partir de las regiones 3’-no traducidas, de cada uno de los diferentes genes de MHC, donde las secuencias son altamente específicas para cada gen de MHC (Tabla 1) (13, 21, 22). Nosotros hemos demostrado previamente que estos primers amplifican los productos PCR de 623, 655 y 609 bp, respectivamente, para ARNm de MHC de Tipo I, IIa y IIb (13, 21).
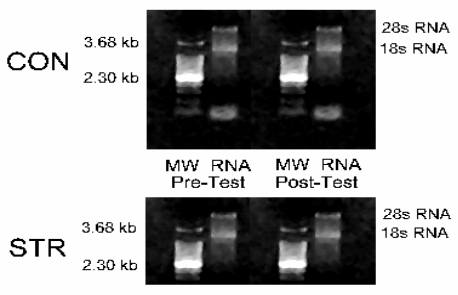
Figura 1. Electroforesis del ARNm total a partir de la homogenización de
una biopsia muscular pre y post-evaluación de los sujetos de los grupos CON y
STR, usada para el procedimiento de PCR cuantitativa. Las abreviaciones son
las siguientes: MW =marcador de peso molecular EcoR-1; 28S ARNr = 28S ARN
ribosomal; 18S ARNr = 18S ARN ribosomal.
Diseño de Patrones de Control de ADN Interno para PCR Cuantitativa
Un patrón de ADN interno fue construido por medio de una técnica de extensión y amplificación de superposición de oligonucleótidos con una temperatura del primer annealing de 55 ºC (5, 23). El patrón de control uso el primer común como su 5’-oligonucleótido y contenía las tres secuencias de isoformas específicas de MHC como su 3’-oligonucleótido. La “secuencia no común” particular no relacionada, originada de un tramo de la región de codificación del gen de la gliceraldehido-3-fosfato dehidrogenasa (GAPDH), que tiene poca o ninguna homología de secuencia con cualquiera de los genes de MHC relevantes.

Tabla 1. Primers de ARN 3’-oligonucleotídicos para la amplificación PCR.. *La muestra
de ADNc es del tamaño del producto de ARNm de MHC de la amplificación PCR en
pares de bases (bp). * El fragmento de control es del tamaño del patrón de
control de ADN interno (bp) co-amplificado con la muestra de ADNc durante la
amplificación PCR, usando los mismos primers del mismo sentido ascendente y en
sentido opuesto descendente.
Clonación de los Patrones de Control de ADN Interno para PCR Cuantitativa
El patrón de control de ADN interno, generado a partir de la PCR fue clonado dentro de un vector de transcripción modificado. Antes de la clonación, fue construido un vector T cortando el vector GEMp-5Zf(+) (Promega, Madison, WI) con la endonucleasa de restricción EcoR-V y adicionando una timina 3’ a ambos extremos. El fragmento de control fue ligado dentro del vector con una ligasa ADN T4 y luego clonado dentro del vector Vector T GEMp (Promega, Madison, WI); un vector en el cual los promotores de la polimerasa ARN T7 y SP6 lindaron con una región de clonación múltiple dentro de la región de codificación alfa-peptídica del gen de la β-galactosidasa.
En este punto, el vector fue transformado en ADN 1 x 108 cfu/μg de bacterias E. Coli JM 109 competentes (Promega, Madison, WI) desparramando alícuotas de la reacción de ligazón en platos de agar que contenían caldo Luria (LB) y ampicilina [(AMP) Sigma Chemical Co., St. Louis, MO] y las células crecieron por una noche a 37 °C. Fueron elegidas colonias de células al azar, cultivadas usando LB y AMP, y el producto PCR del patrón de control interno fue luego purificado por posibles contaminantes como primers dímeros y primers de amplificación, con el Sistema de Purificación de ADN Wizard Plus Midipreps (Promega, Madison, WI).
Identificación y Cuantificación del Patrón de Control Interno
La identificación de los productos PCR para el patrón de control de ADN interno fue verificada por medio de sus longitudes específicas en un gel de agarosa teñido con bromuro de etidio como resultado de una digestión restringida. La secuencia patrón de control de ADN interno fue cortada del ADN con la enzima endonucleasa de restricción EcorR-I (Promega, Madison, WI) y amplificada con PCR con los oligonucleótidos de ARNm de MHC, respectivos (Tabla 1). Los cuales produjeron fragmentos de 462, 510 y 555 bp, respectivamente de ARNm de MHC de Tipo I, IIa y IIb (Figura 2). La verificación posterior del patrón de control de MHC fue determinada por medio de la secuencia de ADN, usando el Sistema de Secuenciamiento de ADN Silver Sequence (Promega, Madison, WI) de acuerdo a los lineamientos de los fabricantes.
La cantidad del patrón de control de ADN interno fue cuantificada determinando la absorción a OD260. La pureza del fragmento fue luego determinada por medio de la determinación de la tasa de absorción entre OD260/OD280 (5, 13, 21).
Amplificación PCR para PCR cuantitativa
Un picogramo (pg = 10-¹² g) del fragmento de control interno y 200 ng de ADNc fueron adicionados a cada reacción de PCR de 25 μL. Para un control negativo absoluto fue amplificada una reacción PCR separada sin contener ningún ADNc. Específicamente, cada reacción PCR contenía las siguientes mezclas: [amortiguador PCR 10 x, 0.2 μM mezcla NTPd, 1.0 μM de un cóctel que contenía los primers oligonucleotídicos de ARN para MHC en sentido y antisentido y cualquiera de los primers específicos para las isoformas I, IIa o IIb (conseguidas a partir de Ransom Hill Biosciences, Ramona, CA) (Tabla 1), 2 mM de Mg Cl2, 1.0 U/μL de polimerasa ADN Taq (Sigma, St. Louis, MO), y nucleasa libre de H2Od]. Cada reacción PCR fue amplificada con un ciclizador térmico (Bio Rad, Hercules, CA). El perfil de amplificación implicó un paso inicial de desnaturalización a 94 °C por 60 segundos, seguido de por 25 ciclos, donde cada ciclo consistió de una desnaturalización a 94 ºC por 45 segundos, primer annealing a 50 ºC por 60 segundos y una extensión a 72 ºC por 90 segundos y un paso final a 72ºC por 180 segundos (5). El número de ciclos fue optimizado para que la señal amplificada estuviera todavía en la porción lineal de un gráfico con la producción expresada como una función de la absorción a OD260 y el número de ciclos (Figura 3). Para evaluar la confiabilidad entre las amplificaciones, fueron realizadas dos amplificaciones PCR separadas para cada muestra.
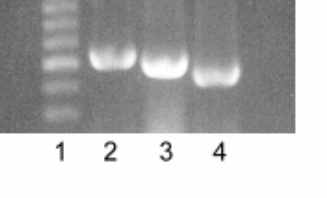
Figura 2. Electroforesis del patrón de control de ADN interno después de la
digestión con la enzima endonucleasa de restricción EcoR-1 y PCR amplificada
con los oligonucleótidos de MHC de Tipo I, IIa, y IIb. Línea 1 = marcador
de ADN MW de 1kb; Línea 2 = patrón de control interno Tipo IIb (555 bp); Línea 3 =
patrón de control interno Tipo IIa (510 bp); Línea 4 = patrón de control
interno Tipo I (462 bp).
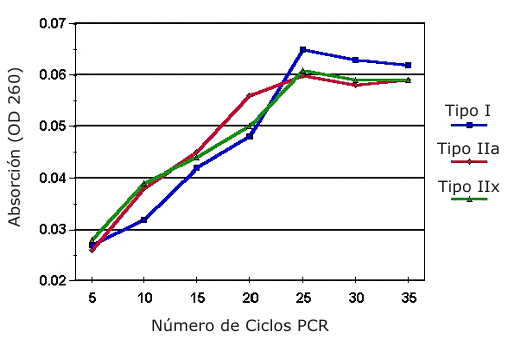
Figura 3. Evaluación de la eficiencia de amplificación con la técnica PCR
cuantitativa. Las curvas de amplificación muestran el número de ciclos
graficados versus la absorción a OD260 para el ARNm de la MHC de Tipo I, IIa,
y IIb co-amplificados con el patrón de control de ADN interno para asegurar que
las mediciones fueron realizadas durante la fase lineal de la amplificación PCR.
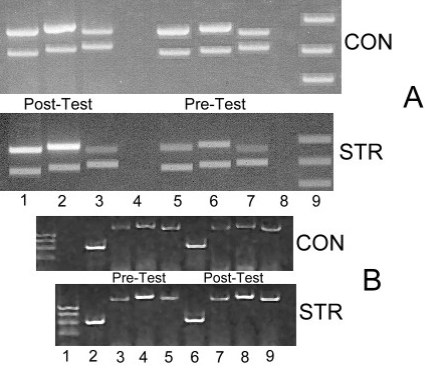
Figura 4. Electroforesis de los productos PCR cualitativos y cuantitativos
de las muestras de biopsias musculares pre y post evaluación para los sujetos
CON y STR. A = PCR Cuantitativa: Los números indican lo siguiente: 1,5 = ARNm
de MHC de Tipo I, 623 bp; 2,6 = ARNm de MHC de Tipo IIa, 655 bp; 3,7= ARNm de MHC
Tipo IIb, 609 bp; 4,8 = control negativo absoluto (ausencia de ADNc);
9= peso molecular del marcador PCR. En los números 1, 2, 3, 5, 6, 7 la menor
banda corresponde al patrón de ADN interno respectivo (Tipo I = 462 bp, IIa =
510 bp, IIb = 555 bp). También los números 1-4 indican pre-evaluación y los
5-8 indican post-evaluación. B = PCR Cualitativa: Los números indican lo
siguiente: 1= peso molecular del marcador PCR, 2,6= alfa-actina [Patrón de
control interno (313 bp)], 3,7 = ARNm de MHC de Tipo I (623 bp), 4,8 = ARNm de
MHC de Tipo IIa (655 bp), 5,9 = ARNm de MHC de Tipo IIb (609 bp). Los números
2-5 indican pre-evaluación y los números 6-9 indican post-evaluación.
Amplificación PCR para PCR Cualitativa
Cuatro mezclas separadas de reacciones PCR se 25 µL [amortuguador PCR 1x, 0.2 µM de mezcla NTPd, 1.0 µM de un cóctel que contenía ambos primers oligonucleotídicos de ARN para MHC en sentido y antisentido, y cualquiera de los tres primers específicos para las isoformas de MHC de Tipo I, IIa y IIb y 0.5 µM de un cóctel que contenía ambos primers oligonucleotídicos de ARN para MHC en sentido y antisentido para a-actina (obtenidos a partir de Ransom Hill Biosciences, Sonoma, CA), 2.5 U/µL de polimerasa ADN Taq (Sigma, St. Louis, MO), 5 µL de ADNc, y nucleasa libre de H2Od] fueron luego amplificadas con un ciclizador térmico (BioRad, Hercules, CA). El perfil de amplificación implico la desnaturalización a 95 ºC por 30 segundos, primer annealing a 55 ºC por 30 segundos, y una extensión a 72 ºC por 1 minuto. Esta secuencia fue repetida por 35 ciclos.
Electroforesis en Gel para PCR Cuantitativa y Cualitativa
Alícuotas (20 μL) de las mezclas de las reacciones PCR cuantitativas y cualitativas fueron sometidas a electroforesis en gel con agarosa al 1,5 % en amortiguador EDTA Tri-acetato 1 x (TAE) (5, 13, 21). Los oligonucleótidos dentro del gel fueron teñidos con bromuro de etidio (presente el el amortiguador TAE a 1 μg/mL), iluminados con un transiluminador UV, y fotografiados con una cámara Polaroid DS 34 (Polaroid, Cambridge, MA). Las fotografías fueron escaneadas (Hewlett Packard 5200C, Palo Alto, CA) y la densidad de las bandas fue determinada usando el software Scion Imaging, expresadas en unidades de densidad arbitrarias (20).
De acuerdo a lineamientos posteriores (3), para la PCR Cuantitativa, solo el volumen del OD de una banda de ADN fue corregido al ambiente local ya que el mismo era directamente proporcional a la cantidad de ADN a través de una amplia extensión. La intensidad de cada banda MHC (expresada en unidades de densidad arbitrarias) fue dividida por la intensidad del patrón de control de ADN interno. El contenido porcentual de ARNm de cada isoforma de MHC fue luego calculado basándose en la fracción del valor corregido de ARNm de MHC específico, relativo a la suma total de las isoformas de ARNm de MHC expresadas en una muestra dada. Por ejemplo, % de ARN de MHC de Tipo IIa = 100 x (Intensidad de banda Tipo IIa/Intensidad de banda de control correspondiente)/ [suma (banda de MHC/banda control) para todas las isoformas de ARNm de MHC] (5).
Aislamiento y Cuantificación de las Proteínas Miofibrilares
Las proteínas miofibrilares, aún permaneciendo en la fase de fenol, dejada luego del procedimiento de aislamiento de ARN total, fueron aisladas con isopropanol, lavadas con hidrocloruro de guanidina 0,3 M y disueltas en SPS al 1 % (24). El contenido de proteínas fue luego determinado espectrofotometricamente basándose en el método de Bradford a una longitud de onda de 595 nm y usando seroalbúmina bovina como patrón (25).
Análisis Estadísticos
Procedimientos de análisis de varianza (ANOVA) separados y a dos vías (Grupo x Evaluación) fueron empleados para determinar diferencias significativas para las siguientes variables de criterio: 1. peso corporal, 2. volumen del muslo, 3. fuerza, 4. porcentaje de ARNm de las isoformas de MHC, y 5. contenido de proteínas miofibrilares. Fueron realizados test-t separados para grupos independientes para determinar diferencias significativas para el porcentaje de cambio de las pre a las post-evaluaciones, con las variables de criterio arriba mencionadas. Adicionalmente, para evaluar la confiabilidad entre las reacciones PCR, fueron realizados test-t apareados para cada sujeto dentro de ambos grupos para las muestras obtenidas por biopsia pre y post entrenamiento. Fue adoptado un nivel de probabilidad de 0.05 para todos los análisis estadísticos.
RESULTADOS
Peso Corporal, Fuerza y Volumen del Muslo
Durante el curso del estudio, no hubo ninguna interacción significativa grupo x evaluación (F (2, 15) =0.14, p>0.05) para el peso corporal (Tabla 2). También, con respecto a los cambios porcentuales en el peso corporal desde las pre a las post evaluaciones, no hubo diferencias significativas (p>0.05) entre los dos grupos.
Como resultado del programa de entrenamiento, fueron encontradas interacciones significativas grupo x evaluación para la fuerza muscular (F (2, 15) =10.44, p =0.007) y volumen del muslo (F (2, 15) =5.50, p =0.02). Los análisis de los efectos principales no revelaron diferencias significativas entre los grupos CON y STR en las pre-evaluaciones para ambas, fuerza muscular (F (2, 15) =1.56, p =0.85) o volumen del muslo (F (2, 15) = 2.13, p =0.23). Al final del estudio para el grupo CON, todavía no había diferencias significativas señaladas para la fuerza o el volumen del muslo, sin embargo, el grupo STR fue significativamente diferente del grupo CON para ambas, fuerza muscular (F (2, 15) =8.33, p =0.005) y volumen del muslo (F (2, 15) =10.65, p =0.007). Con respecto al porcentaje de cambio pre-post entrenamiento para la fuerza muscular, los resultados demostraron incrementos promedio de 2.72 ±1.56 y 48.53 ±8.10 %, respectivamente, para los grupos CON y STR. Para el volumen del muslo, fueron observados incrementos de 0.60 ±2.44 y 11.86 ±8.67 % para los grupos CON y STR, respectivamente. Para la fuerza y el volumen del muslo, los cambios observados para el grupo STR fueron significativamente diferentes con respecto al grupo CON (p<0.05).
Abundancia de Isoformas de ARNm de MHC para la PCR Cuantitativa
Los análisis PCR-RT produjeron productos de amplificación de 623, 655 y 609 bp, respectivamente, para las isofromas de ARNm de MHC de Tipo I, IIa y IIb (bandas superiores, Figura 4). También la co-amplificación del patrón de control de ADN interno produjo productos de amplificación de 462, 510 y 555 bp, respectivamente para MHC de Tipo I, IIa y IIb (bandas inferiores, Figura 4A).
Por la abundancia de ARN de la isoforma de MHC en cada una de las muestras de ambos grupos, fueron encontradas interacciones significativas grupo x evaluación para las isoformas Tipo I (F (2, 15) =13.81, p =0.002), IIa (F (2, 15) =11.61, p =0.001), y IIb (F (2, 15) =13.81, p =0.002). Los análisis de los efectos principales no demostraron diferencias significativas entre los grupos CON y STR en las pre-evaluaciones para la expresión del ARNm de MHC de Tipo I (F (2, 15) =0.614, p =0.55), IIa (F (2, 15) =0.81, p =0.46), y IIb (F (2, 15) =0.21, p =0.89). Al final del estudio, no hubo diferencias significativas en el grupo CON señaladas en la abundancia de ninguna de las tres isoformas de MHC. Sin embargo, después del entrenamiento, se detectaron diferencias significativas para el grupo STR en la abundancia de ARNm de MHC, que fueron significativamente diferentes del grupo CON.
Con respecto al porcentaje de cambio en la abundancia de ARNm desde los pre a los post-test para los dos grupos, los resultados para el grupo CON revelaron incrementos de 1.81 ±3.22 y 2.49 ±5.82 %, y disminuciones de 1.88 ±8.68 %, respectivamente, para las isoformas de MHC de Tipo I, IIa y IIb (Tabla 2). Además, para el grupo STR hubo incrementos promedio de 17.08 ±5.35 y 15.89 ±8.32 %, y disminuciones de 49.69 ±22.38 %, respectivamente, para las isoformas Tipo I, IIa y IIb, que fueron todas significativamente diferentes del grupo CON (p<0.05).
Considerando la confiabilidad intra-ensayo relacionada a la consistencia en la abundancia de ARNm entre las dos amplificaciones PCR dentro de cada grupo, los resultados no demostraron diferencias significativas (p>0.05) para las tres isoformas de MHC. Adicionalmente, el coeficiente de variación intra-ensayo medio para las dos amplificaciones PCR para el grupo CON fue 0.03 ±0.04, 0.02 ±0.04, y 0.03 ±0.03, mientras que los respectivos coeficientes para el grupo STR fueron 0.025 ±0.005, 0.024 ±0.01, y 0.014 ±0.14.
Comparación Cualitativa de la Expresión de las Isoformas de ARNm de MHC
Los patrones de expresión del ARNm de las isoformas de miosina de cadena pesada fueron comparados entre todos los sujetos dentro de los dos grupos. La expresión fue determinada a partir de unidades de densidad arbitraria de los geles teñidos. No fueron detectadas diferencias significativas en las pre-evaluaciones para cualquiera de los dos grupos CON o STR. Sin embargo, en las post-evaluaciones para el grupo STR fueron encontradas diferencias significativas para el ARNm de MHC de las isoformas Tipo I (F (2, 15) =26.43, p =0.002), IIa (F (2, 15) =6.38, p =0.002), y IIb (F (2, 15) =9.60, p =0.001). Con respecto al porcentaje de cambio en la expresión entre las pre y post-evaluaciones para el ARNm de MHC, el grupo CON demostró diferencias aparentes en las isoformas Tipo I, IIa y IIb. Sin embargo, el grupo STR estuvo expresando significativamente más de las isoformas de MHC de Tipo I (19.51 ±8.34%) y Tipo IIa (26.26 ±12.84 %), con una disminución concomitante en el ARNm de la isoforma de Tipo IIb (19.65 ±9.27 %) (Tabla 2 & Figura 4B).
Contenido de Proteínas Miofibrilares
Para el contenido de proteínas miofibrilares, fue encontrada una interacción grupo x evaluación significativa (F (2, 15) =9.74, p =0.006). Los análisis de los efectos principales no revelaron diferencias significativas entre los grupos CON y STR en las pre-evaluaciones (F (2, 15) =2.31, p =0.63). Sin embargo, al final del estudio el contenido de proteínas para el grupo CON no fue significativo, mientras que el grupo STR fue significativamente diferente del grupo CON (F (2, 15)= 11.44, p =0.008).
Con respecto a los cambios porcentuales pre-post entrenamiento para el contenido de proteínas miofibrilares (Tabla 2), los resultados mostraron incrementos promedio de 9.49 ±5.99 y 92.09 ±24.68 %, respectivamente para los grupos CON y STR. Los cambios observados para el grupo STR fueron significativamente diferentes (p<0.05) del grupo CON.
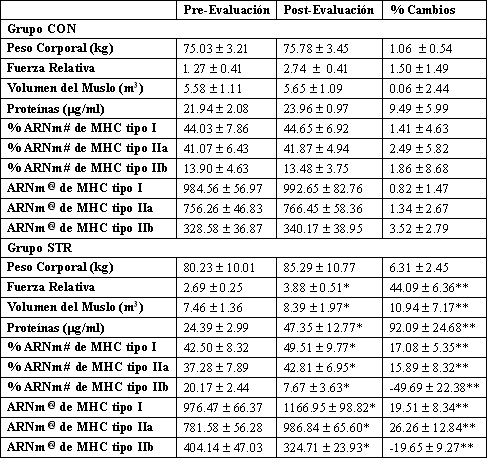
Tabla 2. Medias de los Grupos (±DS) para las variables de Criterio.
*Diferencias significativas con respecto al grupo CON (p < 0.05); **Cambios
significativos desde las pre a las post evaluaciones (p < 0.05); Los
resultados para el % de ARNm de MHC están basados en las densidades
arbitrarias de los productos de MHC amplificados por PCR, relativos al patrón
interno co-amplificado como es ilustrado en la Figura 4. La ecuación para
calcular el % de ARNm de MHC es descrita en la sección de los Métodos y en
otras partes (5). # PCR cuantitativa; @ PCR cualitativa (los números son
expresados en unidades de densidad arbitrarias).
DISCUSIÓN
En términos de la fisiología de la adaptación muscular al entrenamiento de la fuerza, es importante para los fisiólogos del ejercicio entender los mecanismos moleculares en los cuales los genes de MHC pueden ser sensibles a la intensidad de la carga de entrenamiento externa. Como resultado, podemos volvernos más estratégicos para enseñar e implementar programas de entrenamiento de la fuerza que manipulen efectivamente la expresión de los genes de MHC, los cuales juegan un rol significativo en el incremento de la fuerza e hipertrofia muscular. Los estudios que han investigado los efectos del entrenamiento de la fuerza sobre la expresión del gen de MHC en humanos son limitados, y la mayoría de los datos disponibles implican modelos realizados con roedores.
Como puede ser observado en la Figura 4B, la PCR cualitativa solo provee la habilidad para pseudo-cuantificar la expresión de ARNm de la isoforma de MHC a partir de la intensidad de la tinción con bromuro de etidio y normalizada con la alfa-actina como patrón de control externo y positivo. Como resultado, la PCR cualitativa es solo capaz de detectar la presencia o ausencia de ARNm de MHC en una muestra dada y no provee la habilidad para una cuantificación exacta. La tinción con bromuro de etidio con la electroforesis en gel con agarosa y la comparación del ARNm blanco con el ARNm control solo destaca diferencias en las intensidades de tinción, las cuales pueden posiblemente ilustrar diferencias en el contenido de ARNm (Figura 4B). Sin embargo, sin conocer la concentración de la plantilla de iniciación, nuestros intentos para normalizar la expresión comparada con la alfa-actina, fueron problemáticos por dos razones: 1. no hubo cuantificación y estandarización de la concentración inicial de ADNc, y 2. ya que fueron usados diferentes pares de primers para amplificar la MHC comparada con la alfa-actina, no fue posible una completa corrección para la eficiencia de la amplificación PCR. De este modo, este método de PCR cualitativa es estrictamente descriptivo y no se presta para determinar la abundancia de ARNm de la isoforma de MHC como lo hace nuestra técnica cuantitativa (Figura 4).
Basándose en nuestras modificaciones a partir de lineamientos previamente establecidos (5), en este estudio nosotros empleamos un método de PCR específica y competitiva para cuantificar la expresión del ARNm de la isoforma de MHC en humanos. El método PCR descrito aquí implica un gen constitutivamente expresado que es amplificado simultáneamente en la misma reacción como la secuencia de ARNm de MHC blanco, y corregido relativo al gen constitutivo. También, para corregir la variabilidad en la eficiencia de la reacción PCR, fue adicionada a cada reacción una cantidad conocida (1 pg) de un patrón de control de ADN interno y fue amplificada simultáneamente con el ARNm de MHC por medio del uso del mismo set de primers de ARNm de MHC, pero produciendo un producto de diferente tamaño. Como puede ser observado en la Figura 4A, cada reacción PCR resulta en la co-amplificación del ARNm de MHC y el patrón de control de ADN interno. La banda superior corresponde al mayor peso molecular del ARNm de MHC mientras que la banda inferior corresponde al menor peso molecular del fragmento de control de ADN interno. La intensidad de las bandas superiores es indicativa de abundancia de ARNm de MHC específico en la muestra total de ARN, relativa al patrón de control de ADN interno. Este método de PCR-RT competitiva puede estimar exactamente la abundancia de ARNm de MHC en una muestra dada (5), aunque este método solo provee la comparación exacta de la cantidad relativa de expresión de ARNm de cada isoforma de MHC en una muestra dada.
Los resultados de este estudio coinciden con otros estudios que han demostrado que períodos de 9-19 semanas de entrenamiento de la fuerza de alta intensidad fueron efectivos para incrementar la fuerza significativamente (6, 7, 8). Con respecto al volumen del muslo, sin embargo, no hubo cambios significativos (p>0.05) en el grosor del pliegue cutáneo del muslo para el grupo CON (1.56 ±0.04 %) y el grupo STR (1.12 ±0.02 %) (datos no presentados). De este modo, el principal incremento en el volumen de las mediciones vino más probablemente a partir del perímetro del muslo. Es posible que el incremento en el volumen del muslo haya sido debido a incrementos en los niveles de fluido intramuscular y/o intersticial, sin embargo, basándose en los incrementos observados en la fuerza muscular y el contenido de proteínas miofibrilares es más razonable asumir que el incremento del volumen ocurrió como resultado de hipertrofia de la musculatura del muslo. Los presentes resultados ilustran incrementos significativos inducidos por el entrenamiento en las proteínas miofibrilares que coinciden con estudios previos (9, 10). Durante la hipertrofia inducida por el entrenamiento de fuerza, hay una acumulación incrementada de proteínas no contráctiles y contráctiles. La cantidad neta de estas proteínas es un resultado de tasas de síntesis vs. degradación y parece que la síntesis de proteínas miofibrilares puede estar regulada al nivel de traducción o post-traducción (26). Los presentes resultados también demuestran que los niveles de ARNm de MHC en estado estable pre-ejercicio son similares a otras investigaciones que involucraron humanos (11) en las cuales el músculo no ejercitado expresó un mayor porcentaje de la isoforma Tipo I, con la isoforma Tipo IIa expresándose más que la IIb. Nuestros resultados, ilustran que el entrenamiento de la fuerza de alta intensidad parece causar transiciones rápidas a lentas en la expresión del ARNm de la isoforma de MHC que parece ser paralela a las transiciones de las isoformas de proteínas MHC vistas en otros estudios (6, 7, 8). El hecho que nosotros demostramos incrementos en la expresión del ARNm de la isoforma de MHC en comparación con otros estudios que no lo hicieron (11, 12) puede haber sido debido a la mayor intensidad de entrenamiento y/o a la duración del período de entrenamiento del presente estudio. Los resultados del presente estudio también indican un incremento significativo en la abundancia de ARNm de MHC de Tipo I y IIa, luego del entrenamiento acoplado con una disminución concomitante en la abundancia de la isoforma Tipo IIb. La abundancia disminuida en la isoforma Tipo IIb puede haberse debido a la premisa de que el gen IIb es conocido como el “gen rebelde” (27) y es muy sensible al entrenamiento de la fuerza de alta intensidad y a las alteraciones en la carga mecánica y diferentes velocidades de contracción muscular. De este modo, basándose en el modo y la intensidad del estímulo de entrenamiento, y posiblemente el modelo animal usado, el gen Tipo IIb puede tener la habilidad de preferentemente, ya sea incrementar o disminuir su expresión, basándose en los perfiles de reclutamiento de las fibras musculares durante la actividad. Ya que el entrenamiento de la fuerza de alta intensidad implica grandes esfuerzos de contracción pero bajas velocidades de acortamiento de las fibras musculares, las fibras musculares Tipo IIb pueden no ser tan fuertemente reclutadas como las fibras Tipo I y IIa (29). Como resultado, los perfiles de reclutamiento de fibras musculares, relacionados a la intensidad de entrenamiento (carga) y a la velocidad de la contracción, pueden haber jugado un rol para dictar la disminución de la regulación en la expresión del gen Tipo IIb. Como resultado, esto puede implicar que, con respecto al principio de especificidad, la intensidad del entrenamiento de la fuerza puede jugar realmente un rol clave en las diferentes expresiones de los genes de MHC de Tipo I, IIa y IIb. De este modo, los regimenes de entrenamiento deberían ser específicos basándose en el tipo de ejercicio y/o en la participación en diferentes deportes.
El incremento de la plantilla de ARN disponible y la síntesis de proteínas en el músculo durante la hipertrofia puede ser el reflejo de alteraciones en la eficiencia transcripcional, la capacidad transcripcional y/o la estabilidad del ARNm. Los resultados de este estudio indican que los incrementos en la abundancia de ARNm de MHC de Tipo I y IIa ocurren concomitantemente con incrementos en el contenido de proteínas miofibrilares. De este modo, puede existir una posible relación entre el aumento de la regulación de la expresión de los genes de MHC Tipo I y IIa y el entrenamiento de la fuerza de alta intensidad que es necesario para apoyar los incrementos inducidos por el entrenamiento en la fuerza y el volumen del muslo, observado en este estudio.
Debería ser destacado, sin embargo, que el método de cuantificación de ARNm empleado en este estudio solo refleja niveles de ARNm en estado estable al tiempo de las biopsias. Cuando se interpreta la expresión del ARNm de la isoforma de MHC, durante períodos de inactividad física, se demuestra frecuentemente que la expresión de ARNm de MHC y de proteínas ocurre en la misma dirección (26). Sin embargo, durante períodos de entrenamiento e hipertrofia muscular, parece ocurrir un desequilibrio entre el contenido relativo de ARNm de MHC y los niveles de proteínas, los cuales pueden estar rejlejando diferencias en la coordinación entre el aumento y la disminución de la regulación de la transcripción de MHC y de sus isoformas de proteínas (29). Este desequilibrio entre la actividad pre-traducción y de traducción puede ser debido a la naturaleza transitoria (0-4 horas post-ejercicio) de la expresión de los genes de MHC en comparación a la expresión de las proteínas MHC (3-36 horas post-ejercicio) (26). Como resultado de la naturaleza transitoria de la expresión de ARNm, puede ser requerido más de un solo punto en el tiempo de determinación del ARNm para confirmar interpretaciones absolutas de los sitios de regulación de la expresión del gen MHC.
Referencias
1. Mahdavi V, Izumo S, Nadal-Ginard B (1987). Developmental and hormonal regulation of sarcomeric myosin heavy gene family. Circul Res; 60: 804-814
2. Perisamy M, Gregory P, Martin B, Stirewalt W (1989). Regulation of myosin heavy chain gene expression during skeletal muscle hypertrophy. Biochem J; 257: 691-698
3. Jankala H, Harjola V, Petersen N, Harkonen M (1997). Myosin heavy chain mRNA transform to faster isoforms in immobilized skeletal muscle: a quantitative PCR study. J Appl Physiol; 83: 977-982
4. Booth F, Kirby C (1992). Changes in skeletal muscle gene expression consequent to altered weight bearing. Am J Physiol; 262: R329-R332
5. Wright C, Haddad F, Qin A, Baldwin K (1997). Analysis of myosin heavy chain mRNA expression by RT-PCR. J Appl Physiol; 83: 1389-1396
6. Adams G, Hather B, Baldwin K, Dudley G (1993). Skeletal muscle myosin heavy chain composition and resistance training. J Appl Physiol; 74: 911-915
7. Jurimae J, Abernethy P, Blake K, McEniery M (1996). Changes in the myosin heavy chain isoform profile of the triceps brachii muscle following 12 weeks of resistance training. Eur J Appl Physiol; 74: 287-292
8. Staron R, Karapondo D, Kraemer W, Fry A, Gordon S, Falkel J, Hagerman F, Hikida R (1994). Skeletal muscle adaptations during early phase of heavy-resistance training in men and women. J Appl Physiol; 76: 1247-1255
9. Chesley A, MacDougall D, Tarnopolsky M, Atkinson S, Smith K (1992). Changes in human muscle protin synthesis after resistance exercise. J Appl Physiol; 73: 1383-1388
10. Yarasheski K, Zachwieja J, Bier D (1993). Acute effects of resistance exercise on muscle protein synthesis rate in young and elderly men and women. Am J Physiol; 265: E210-E214
11. Welle S, Bhatt K, Thornton C (1999). Stimulation of myofibrillar synthesis by exercise is mediated by more efficient translation of mRNA. J Appl Physiol; ; 86: 1220-1225
12. Hortabayagi T, Dempsey L, Fraser D, Zheng D, Hamilton G, Lambert J (2000). Changes in muscle strength, muscle fiber size and miofibrillar gene expression after immobilization and retraining in humans. J Physiol; 524: 293-304
13. Willoughby D, Pelsue S (1998). Muscle strength and qualitative myosin heavy chain isoform mRNA expression in the elderly after moderate- and high-intensity weight training. J Aging Phys Act; 6: 327-339
14. Stedman H, Satyapriya S (1988). Molecular genetics in basic myology: A rapidly evolving perspective. Muscle Nerve; 11: 668-682
15. Willoughby D (1993). The effects of weight training programs involving periodization and partially-equated volumes on upper- and lower-body strength. J Strength Conditioning Res; 7: 2-8
16. Willoughby D, Simpson S (1996). The effects of combined electromyostimulation and dynamic muscular contractions on the strength on college basketball players. J Strength Conditioning Res; 10: 34-39
17. Andersen P, Saltin B (1985). Maximal perfusion of skeletal muscle in man. J Physiol; 366: 233-249
18. Berger R (1962). Optimum repetitions for the development of strength. Res Quart Exer Sport; 33: 334-338
19. Fleck S. Kraemer W (1999). . Designing Resistance Training Programs (3rd ed.). Champaingn, IL: Human Kinetics
20. Willoughby D, Priest J, Jennings R (1940). . Myosin heavy chain isoform and ubiquitin protease mRNA expression after passive leg cycling in persons with spinal cord injury. Arch Phys Med Rehab; In Press 2003
21. Willoughby D, Priest J, Jennings R (2003). Myosin heavy chain isoform and ubiquitin protease mRNA expression after passive leg cycling in persons with spinal cord injury. Arch Phys Med Rehab; In Press
22. Ennion S, Sant'Ana Pereira J, Sargent A, Young A, Goldspink G (1995). Characterization of human skeletal muscle fibers according to the myosin heavy chains they express. J Mus Res Cell Mot; 16: 35-43
23. Wang A, Doyle M, Mark D (1989). Quantitation of mRNA by the polymerase chain reaction. Biochemistry; 86: 9717-9721
24. Chomczynski P (1993). A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques; 15: 532-537
25. Willoughby D. Barnes W. Ingalls C. Smith S (1996). Effects of the beta2-agonist clenbuterol and exercise training on muscle protein and performance in frogs. J Strength Conditioning Res; 10: 228-233
26. Booth F. Tseng B. Fluck M (1998). Carson J. Molecular and cellular adaptations of muscle in response to physical training. Acta Physiol Scand; 162: 343-350
27. 27. Goldspink G. Scutt A. Martindale J. Jaenicke T. Turay L. Gerlach G (1991). Stretch and force generation induce rapid hypertrophy and myosin isoform gene switching in adult skeletal muscle. Biochem Soc Trans; 19: 368-373
28. Caiozzo V (1996). Haddad F. Baker M. Baldwin K. Influence of mechanical loading on myosin heavy-chain protein and mRNA isoform expression. J Appl Physiol; 80: 1503-1512
29. Staron R. Hikida R. Hagerman F. Dudley G (1984). Murray T. Human skeletal muscle fiber type adaptability to various workloads. J Histochem Cytochem; 32: 146-152